Prévision de défaut du paiement avec Xgboost
Dans ce post, nous allons entraîner et régler les hyperparamètres du modèle XGBoost. On va utiliser le package tidymodels, qui se compose de plusieurs library, un workflow pour entrainer un modèle de classification:
- recipes : framework pour le Preprocessing
- rsample : Cross Validation et sample
- parsnip : framework de train des Machines Learnings
- tune : Tuning des hyperparamètres
- yardstick : evaluation sur les données Test
Ce projet consiste à construire la meilleure fonction score possible sur une base de données classique en machine learning : la base Credit Data disponible dane le package modeldata.
Load packages necessaires
Load les library
library(tidymodels)
library(dplyr)
library(modeldata)
for EDA
library(summarytools)
library(skimr)
library(DataExplorer)
Helper packages
library(modeldata) # for Data score
library(vip)
Parallel processing
library(doParallel)Load les données
set.seed(123)
data("credit_data", package = "modeldata")
credit_data %>% glimpse()# Rows: 4,454
# Columns: 14
# $ Status <fct> good, good, bad, good, good, good, good, good, good, bad, go~
# $ Seniority <int> 9, 17, 10, 0, 0, 1, 29, 9, 0, 0, 6, 7, 8, 19, 0, 0, 15, 33, ~
# $ Home <fct> rent, rent, owner, rent, rent, owner, owner, parents, owner,~
# $ Time <int> 60, 60, 36, 60, 36, 60, 60, 12, 60, 48, 48, 36, 60, 36, 18, ~
# $ Age <int> 30, 58, 46, 24, 26, 36, 44, 27, 32, 41, 34, 29, 30, 37, 21, ~
# $ Marital <fct> married, widow, married, single, single, married, married, s~
# $ Records <fct> no, no, yes, no, no, no, no, no, no, no, no, no, no, no, yes~
# $ Job <fct> freelance, fixed, freelance, fixed, fixed, fixed, fixed, fix~
# $ Expenses <int> 73, 48, 90, 63, 46, 75, 75, 35, 90, 90, 60, 60, 75, 75, 35, ~
# $ Income <int> 129, 131, 200, 182, 107, 214, 125, 80, 107, 80, 125, 121, 19~
# $ Assets <int> 0, 0, 3000, 2500, 0, 3500, 10000, 0, 15000, 0, 4000, 3000, 5~
# $ Debt <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 2500, 260, 0, 0, 0, 2000~
# $ Amount <int> 800, 1000, 2000, 900, 310, 650, 1600, 200, 1200, 1200, 1150,~
# $ Price <int> 846, 1658, 2985, 1325, 910, 1645, 1800, 1093, 1957, 1468, 15~Exploaration des données - EDA avec skimr
skim(credit_data)| Name | credit_data |
| Number of rows | 4454 |
| Number of columns | 14 |
| _______________________ | |
| Column type frequency: | |
| factor | 5 |
| numeric | 9 |
| ________________________ | |
| Group variables | None |
Variable type: factor
| skim_variable | n_missing | complete_rate | ordered | n_unique | top_counts |
|---|---|---|---|---|---|
| Status | 0 | 1 | FALSE | 2 | goo: 3200, bad: 1254 |
| Home | 6 | 1 | FALSE | 6 | own: 2107, ren: 973, par: 783, oth: 319 |
| Marital | 1 | 1 | FALSE | 5 | mar: 3241, sin: 977, sep: 130, wid: 67 |
| Records | 0 | 1 | FALSE | 2 | no: 3681, yes: 773 |
| Job | 2 | 1 | FALSE | 4 | fix: 2805, fre: 1024, par: 452, oth: 171 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| Seniority | 0 | 1.00 | 7.99 | 8.17 | 0 | 2.00 | 5 | 12.0 | 48 | ▇▃▁▁▁ |
| Time | 0 | 1.00 | 46.44 | 14.66 | 6 | 36.00 | 48 | 60.0 | 72 | ▁▂▅▃▇ |
| Age | 0 | 1.00 | 37.08 | 10.98 | 18 | 28.00 | 36 | 45.0 | 68 | ▆▇▆▃▁ |
| Expenses | 0 | 1.00 | 55.57 | 19.52 | 35 | 35.00 | 51 | 72.0 | 180 | ▇▃▁▁▁ |
| Income | 381 | 0.91 | 141.69 | 80.75 | 6 | 90.00 | 125 | 170.0 | 959 | ▇▂▁▁▁ |
| Assets | 47 | 0.99 | 5403.98 | 11574.42 | 0 | 0.00 | 3000 | 6000.0 | 300000 | ▇▁▁▁▁ |
| Debt | 18 | 1.00 | 343.03 | 1245.99 | 0 | 0.00 | 0 | 0.0 | 30000 | ▇▁▁▁▁ |
| Amount | 0 | 1.00 | 1038.92 | 474.55 | 100 | 700.00 | 1000 | 1300.0 | 5000 | ▇▆▁▁▁ |
| Price | 0 | 1.00 | 1462.78 | 628.13 | 105 | 1117.25 | 1400 | 1691.5 | 11140 | ▇▁▁▁▁ |
Splitting – la répartition des données
Maintenant, on divise les données en données de validation et de test. Les données de validation sont utilisées pour l’entrainement du modèle et l’ajustement des hyperparamètres. Une fois le modèle construit, il peut être évalué par rapport aux données test pour évaluer la qualité et la précision du nouveau modèle.
split <- initial_split(credit_data, prop = 0.8, strata = Status)
split# <Analysis/Assess/Total>
# <3563/891/4454>train_data <- training(split)
test_data <- testing(split)Preprocessing
Le prétraitement modifie les données pour rendre notre modèle plus prédictif et le processus de formation moins intensif. De nombreux modèles nécessitent un prétraitement minutieux et exhaustif des variables pour produire des prévisions précises. XGBoost, cependant, est robuste contre les données fortement asymétriques et/ou corrélées. Néanmoins, nous pouvons encore bénéficier d’un certain prétraitement
Dans tidymodels, nous utilisons recipes pour définir ces étapes de prétraitement. Le preprocessing a été déini au départ pour entrainer le modéle LightGBM
recipe_credit <- train_data %>%
recipe(Status ~ .) %>%
step_unknown(all_nominal(), -Status) %>%
step_medianimpute(all_numeric()) %>%
step_other(all_nominal(), -Status, other = "infrequent_combined") %>%
step_novel(all_nominal(), -Status, new_level = "unrecorded_observation") %>%
step_dummy(all_nominal(), -Status, one_hot = TRUE) %>%
step_mutate(
ratio_expense_income = Expenses / (Income + 0.001),
ratio_asset_income = Assets / (Income + 0.001),
ratio_debt_asset = Debt / (Assets + 0.001),
ratio_debt_income = Debt / (Income + 0.001),
ratio_amount_price = Amount / (Price + 0.010)
)
# Add upsmpling
# step_upsample(Status, over_ratio = tune())
recipe_credit# Data Recipe
#
# Inputs:
#
# role #variables
# outcome 1
# predictor 13
#
# Operations:
#
# Unknown factor level assignment for all_nominal(), -Status
# Median Imputation for all_numeric()
# Collapsing factor levels for all_nominal(), -Status
# Novel factor level assignment for all_nominal(), -Status
# Dummy variables from all_nominal(), -Status
# Variable mutation for ratio_expense_income, ratio_asset_income, ratio_debt_asset, ratio_debt_income, ratio_amount_priceSplitting pour la validation croisée
cv_fold <- vfold_cv(train_data, v = 5, strata = Status)
cv_fold# # 5-fold cross-validation using stratification
# # A tibble: 5 x 2
# splits id
# <list> <chr>
# 1 <split [2850/713]> Fold1
# 2 <split [2850/713]> Fold2
# 3 <split [2850/713]> Fold3
# 4 <split [2851/712]> Fold4
# 5 <split [2851/712]> Fold5Specifications du modéle XGboost
On utilise le package parsnip pour définir la spécification du modèle. Parsnip permet d’accéder à plusieurs packages d’apprentissage automatique les 3 étapes:
1- définir le type du modèle à entrainer
2- Décider quel moteur (engine) de calcul à utiliser
3- définir le mode “classification”
xgb_spec <- boost_tree(
trees = tune(),
tree_depth = tune(), min_n = tune(),
loss_reduction = tune(),
sample_size = tune(), mtry = tune(),
learn_rate = tune()
) %>%
set_engine("xgboost") %>%
set_mode("classification")
xgb_spec %>% translate()# Boosted Tree Model Specification (classification)
#
# Main Arguments:
# mtry = tune()
# trees = tune()
# min_n = tune()
# tree_depth = tune()
# learn_rate = tune()
# loss_reduction = tune()
# sample_size = tune()
#
# Computational engine: xgboost
#
# Model fit template:
# parsnip::xgb_train(x = missing_arg(), y = missing_arg(), colsample_bytree = tune(),
# nrounds = tune(), min_child_weight = tune(), max_depth = tune(),
# eta = tune(), gamma = tune(), subsample = tune(), nthread = 1,
# verbose = 0)Grid specification
On va utiliser grid_latin_hypercube afin que nous puissions couvrir l’espace des hyperparamètres le mieux possible
xgb_grid <- grid_latin_hypercube(
trees(),
tree_depth(),
min_n(),
loss_reduction(),
sample_size = sample_prop(),
finalize(mtry(), train_data),
learn_rate(),
size = 50
)
xgb_grid# # A tibble: 50 x 7
# trees tree_depth min_n loss_reduction sample_size mtry learn_rate
# <int> <int> <int> <dbl> <dbl> <int> <dbl>
# 1 32 1 33 0.00122 0.128 12 7.92e- 5
# 2 1667 6 23 0.00000428 0.754 14 4.51e- 5
# 3 1846 9 7 0.0000000700 0.513 2 4.77e- 2
# 4 697 7 11 0.000000112 0.651 4 5.39e- 3
# 5 235 13 21 0.00000260 0.240 5 1.19e- 6
# 6 785 13 19 0.0000000144 0.211 12 1.70e- 3
# 7 1336 11 26 0.0852 0.270 9 5.46e- 8
# 8 1185 2 2 0.0000319 0.354 12 2.47e-10
# 9 1623 1 29 0.0000566 0.538 8 6.50e-10
# 10 181 7 15 0.0000170 0.967 10 4.41e- 6
# # ... with 40 more rowsDéfinir workflow
xgb_wf <- workflow() %>%
add_recipe(recipe_credit) %>%
add_model(xgb_spec)
xgb_wf# == Workflow ====================================================================
# Preprocessor: Recipe
# Model: boost_tree()
#
# -- Preprocessor ----------------------------------------------------------------
# 6 Recipe Steps
#
# * step_unknown()
# * step_medianimpute()
# * step_other()
# * step_novel()
# * step_dummy()
# * step_mutate()
#
# -- Model -----------------------------------------------------------------------
# Boosted Tree Model Specification (classification)
#
# Main Arguments:
# mtry = tune()
# trees = tune()
# min_n = tune()
# tree_depth = tune()
# learn_rate = tune()
# loss_reduction = tune()
# sample_size = tune()
#
# Computational engine: xgboostTune des hyperparamètres
Pour accélérer le calcul, on utilise paralell processing. Pour en savoir plus https://curso-r.github.io/treesnip/articles/parallel-processing.html
all_cores <- parallel::detectCores(logical = FALSE)
registerDoParallel(cores = all_cores)
set.seed(452)
library(tictoc)
cat("Nombre de cores :", all_cores)# Nombre de cores : 4Le tuning prends un peu de temps pour évaluer l’ensemble des paramétres.
tic()
xgb_tune <- tune_grid(
xgb_wf,
resamples = cv_fold,
# metrics = metric_set(roc_auc, j_index, sens, spec),
control = control_grid(verbose = FALSE, save_pred = TRUE)
)
toc()# 108.1 sec elapsedxgb_tune
Evaluer le Résultat
On va sortir les paramètres de tous ces modèles.
DT::datatable(xgb_tune %>% collect_metrics())ROC curve vs gain curve
library(patchwork)
p1 <- xgb_tune %>%
collect_predictions() %>%
group_by(.config) %>%
roc_curve(Status, .pred_bad) %>%
autoplot() + theme(legend.position = "none")
p2 <- xgb_tune %>%
collect_predictions() %>%
group_by(.config) %>%
gain_curve(Status, .pred_bad) %>%
autoplot() + theme(legend.position = "none")
# p1 / p2
p1 + p2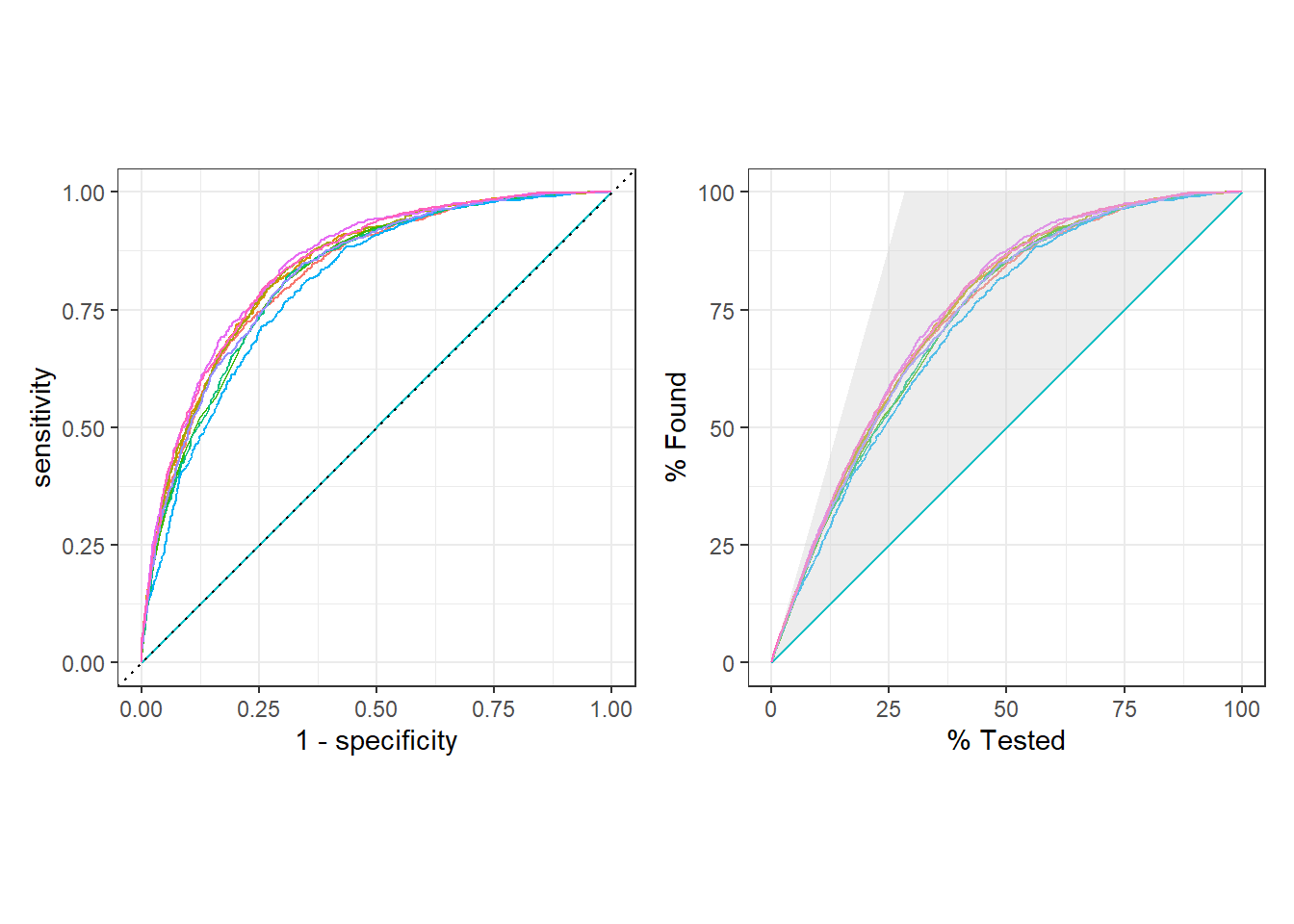
Quels sont les combinaisons de paramètres les plus performants?
Et aprés, finaliser le modèle XGBoost avec les meilleurs paramètres.
xgb_best <- select_best(xgb_tune, "roc_auc")
xgb_final_wf <- finalize_workflow(xgb_wf, xgb_best)
xgb_best# # A tibble: 1 x 8
# mtry trees min_n tree_depth learn_rate loss_reduction sample_size .config
# <int> <int> <int> <int> <dbl> <dbl> <dbl> <chr>
# 1 28 1974 12 10 0.00187 0.0458 0.798 Preprocess~xgb_tune %>%
collect_metrics(parameters = xgb_best)# # A tibble: 20 x 13
# mtry trees min_n tree_depth learn_rate loss_reduction sample_size .metric
# <int> <int> <int> <int> <dbl> <dbl> <dbl> <chr>
# 1 3 1181 40 2 8.02e- 5 4.49e- 4 0.954 accuracy
# 2 3 1181 40 2 8.02e- 5 4.49e- 4 0.954 roc_auc
# 3 6 883 24 4 1.24e- 5 4.81e- 7 0.875 accuracy
# 4 6 883 24 4 1.24e- 5 4.81e- 7 0.875 roc_auc
# 5 10 287 5 14 4.05e- 4 5.58e- 5 0.166 accuracy
# 6 10 287 5 14 4.05e- 4 5.58e- 5 0.166 roc_auc
# 7 14 1337 19 13 2.24e- 9 7.30e- 9 0.283 accuracy
# 8 14 1337 19 13 2.24e- 9 7.30e- 9 0.283 roc_auc
# 9 17 1610 26 11 1.70e- 8 7.21e- 3 0.485 accuracy
# 10 17 1610 26 11 1.70e- 8 7.21e- 3 0.485 roc_auc
# 11 20 41 30 3 1.28e-10 4.59e- 1 0.273 accuracy
# 12 20 41 30 3 1.28e-10 4.59e- 1 0.273 roc_auc
# 13 22 507 33 7 1.70e- 7 6.73e+ 0 0.372 accuracy
# 14 22 507 33 7 1.70e- 7 6.73e+ 0 0.372 roc_auc
# 15 27 1421 8 6 2.17e- 6 1.79e- 7 0.669 accuracy
# 16 27 1421 8 6 2.17e- 6 1.79e- 7 0.669 roc_auc
# 17 28 1974 12 10 1.87e- 3 4.58e- 2 0.798 accuracy
# 18 28 1974 12 10 1.87e- 3 4.58e- 2 0.798 roc_auc
# 19 31 740 14 9 2.20e- 2 1.97e-10 0.585 accuracy
# 20 31 740 14 9 2.20e- 2 1.97e-10 0.585 roc_auc
# # ... with 5 more variables: .estimator <chr>, mean <dbl>, n <int>,
# # std_err <dbl>, .config <chr>Déterminer les paramètres les plus importants
library(vip)
xgb_final_wf %>%
fit(data = train_data) %>%
pull_workflow_fit() %>%
vip(geom = "point")# [22:34:34] WARNING: amalgamation/../src/learner.cc:1061: Starting in XGBoost 1.3.0, the default evaluation metric used with the objective 'binary:logistic' was changed from 'error' to 'logloss'. Explicitly set eval_metric if you'd like to restore the old behavior.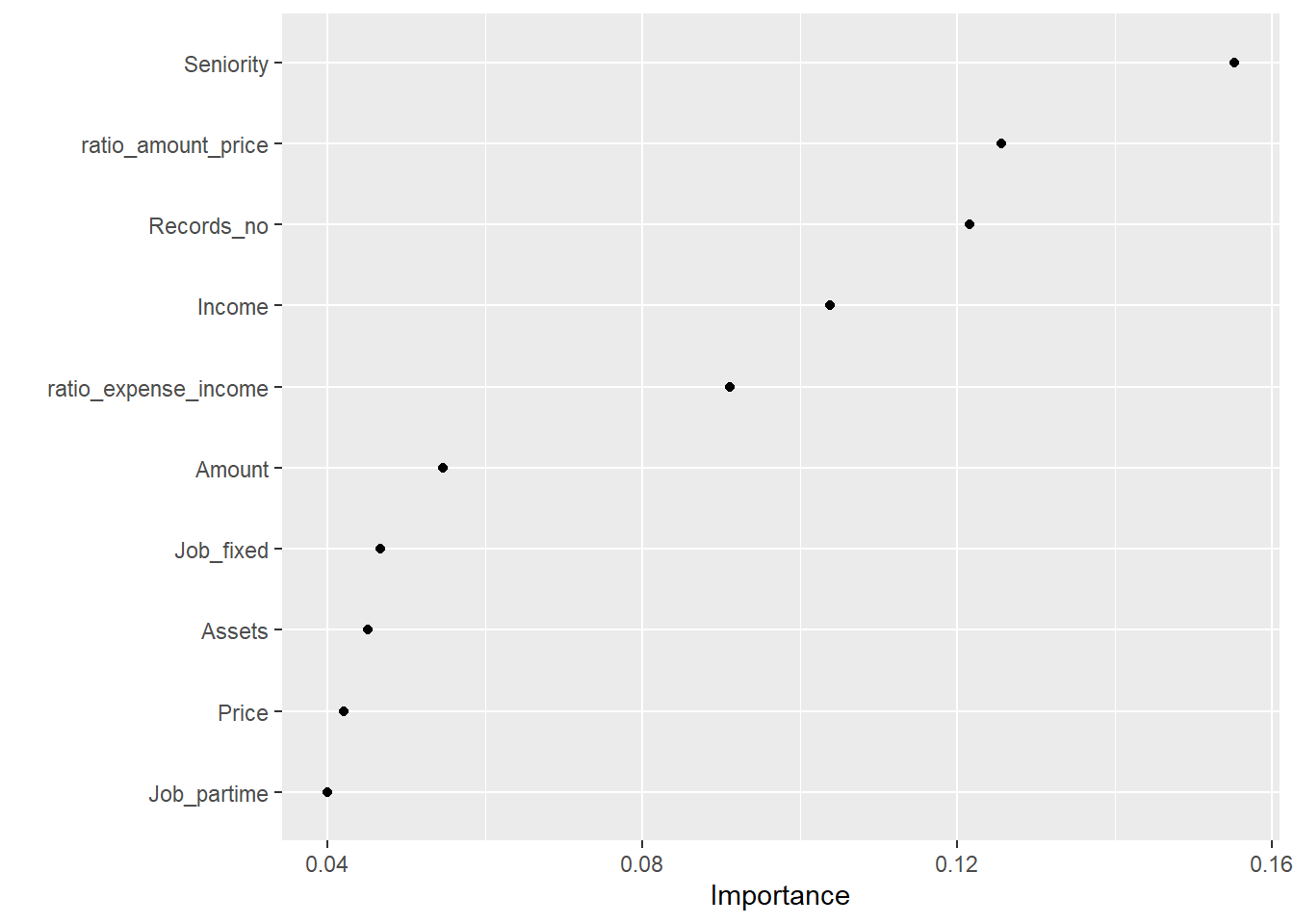
last_fit() pour adapter notre modèle une dernière fois sur les données de validation et évaluer notre modèle sur les données de tests
xgb_final <- xgb_final_wf %>%
last_fit(split, metrics = metric_set(roc_auc, j_index, sens, spec, accuracy))
xgb_final %>% collect_metrics()# # A tibble: 5 x 4
# .metric .estimator .estimate .config
# <chr> <chr> <dbl> <chr>
# 1 j_index binary 0.410 Preprocessor1_Model1
# 2 sens binary 0.482 Preprocessor1_Model1
# 3 spec binary 0.928 Preprocessor1_Model1
# 4 accuracy binary 0.802 Preprocessor1_Model1
# 5 roc_auc binary 0.847 Preprocessor1_Model1La courbe ROC nous donnera une idée de la performance de notre modèle avec les données tests. Vous devriez savoir que si AUC (Area Under Curve) est proche de 50%, alors le modèle est aussi bon qu’un sélection aléatoire; par contre, si AUC est proche de 100%, vous avez un «modèle parfait»
xgb_final %>%
collect_predictions() %>%
roc_curve(Status, .pred_bad) %>%
autoplot()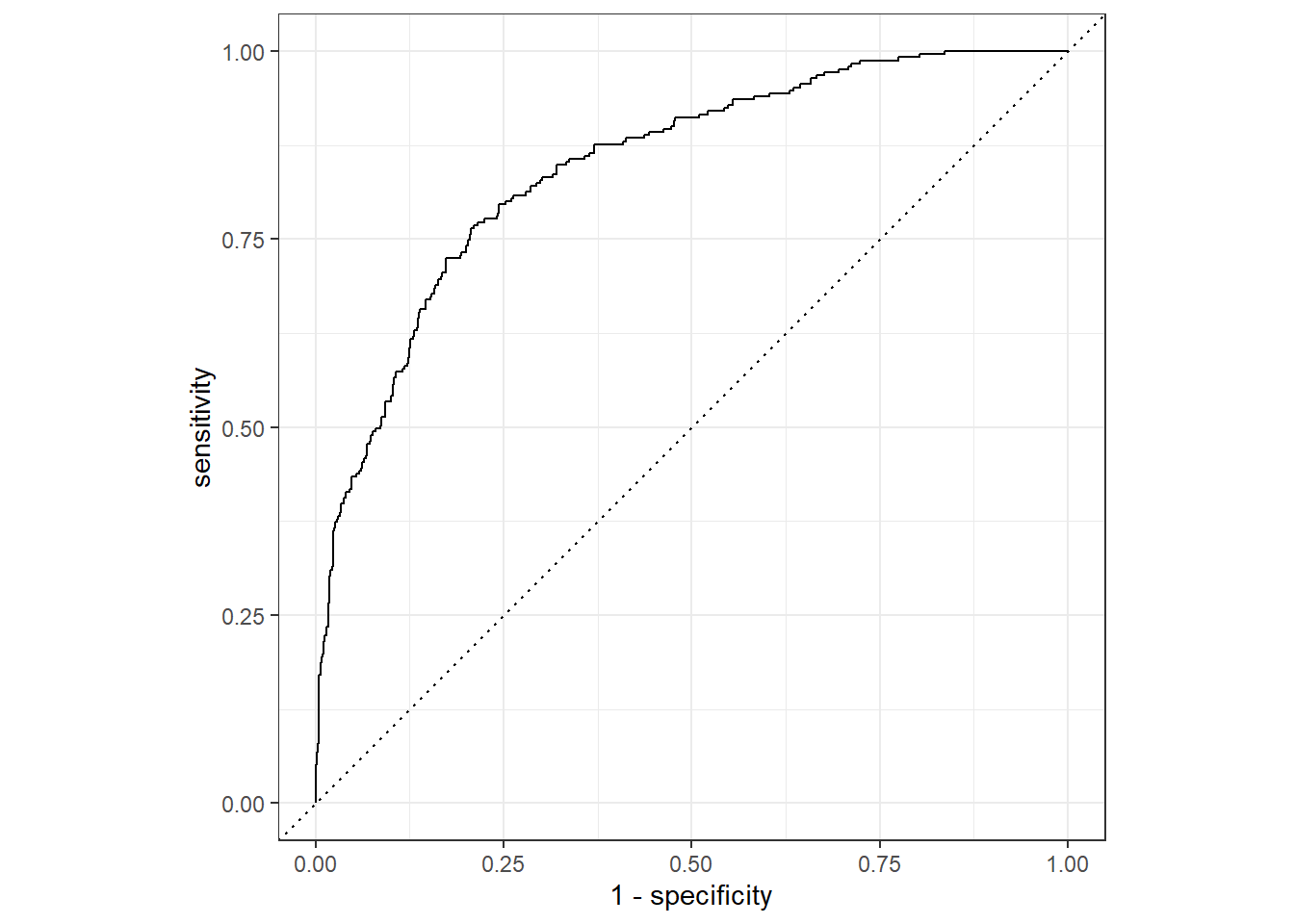
xgb_final %>%
collect_predictions() %>%
roc_curve(Status, .pred_bad) %>%
ggplot(aes(x = 1 - specificity, y = sensitivity)) +
geom_path(lwd = 1.5, alpha = 0.8, color = "#421269") +
geom_abline(lty = 3) +
geom_hline(yintercept = 0.42) +
geom_vline(xintercept = 1 - 0.9186228) +
coord_equal() +
# scale_color_viridis_d(option = "plasma", end = .6)+
theme_bw()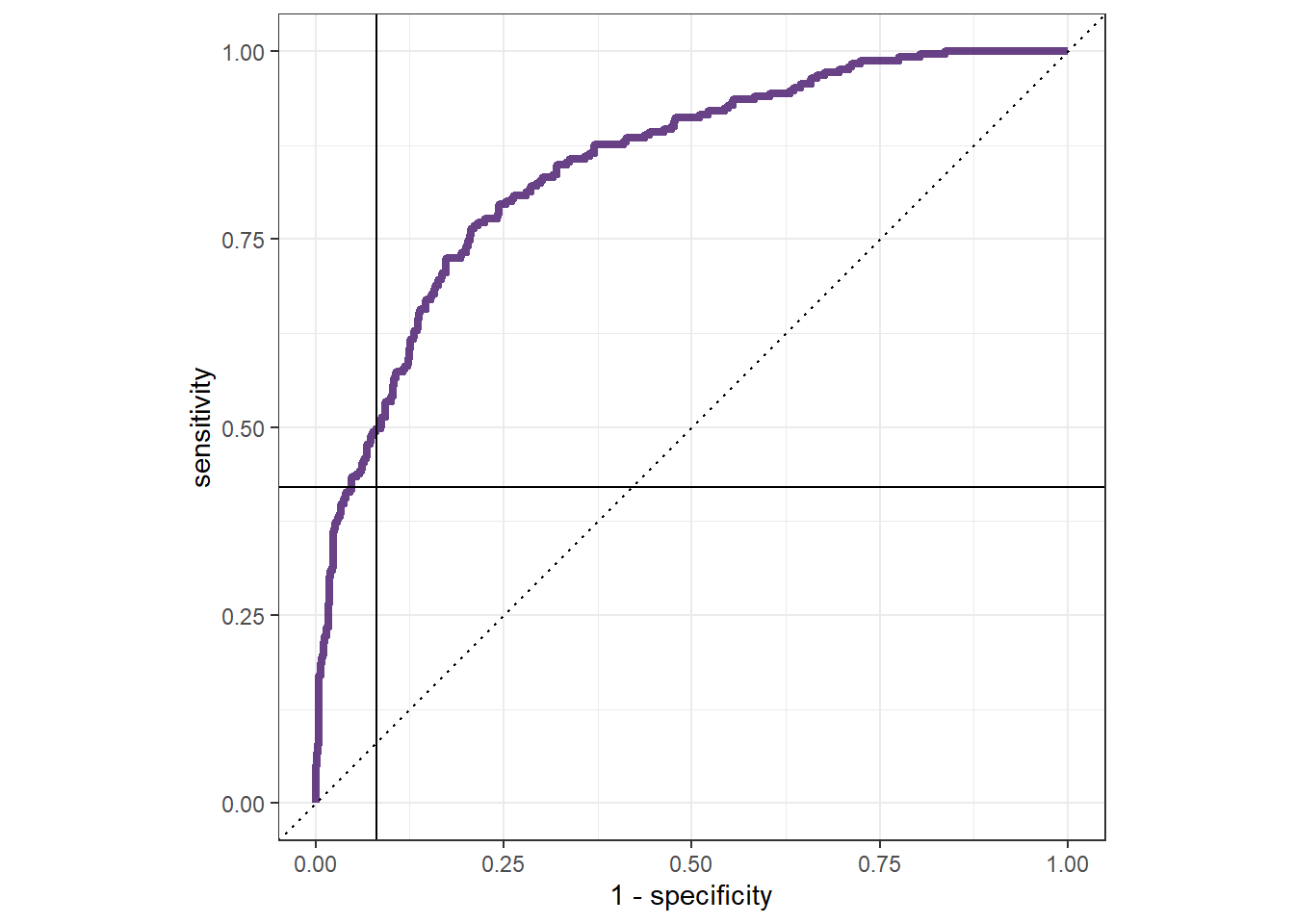
Analyse de la densité pour chaque classe vs prévision, ce graphe permet de fournir des informations visuelles sur l’asymétrie, la distribution, et la qualité de prévision de notre modèle.
xgb_auc <- xgb_final %>% collect_predictions()
h1 <- xgb_auc %>%
ggplot() +
geom_density(aes(x = .pred_bad, fill = Status), alpha = 0.5) +
theme_bw()
h2 <- xgb_auc %>%
ggplot() +
geom_density(aes(x = .pred_good, fill = Status), alpha = 0.5) +
theme_bw()
h1 / h2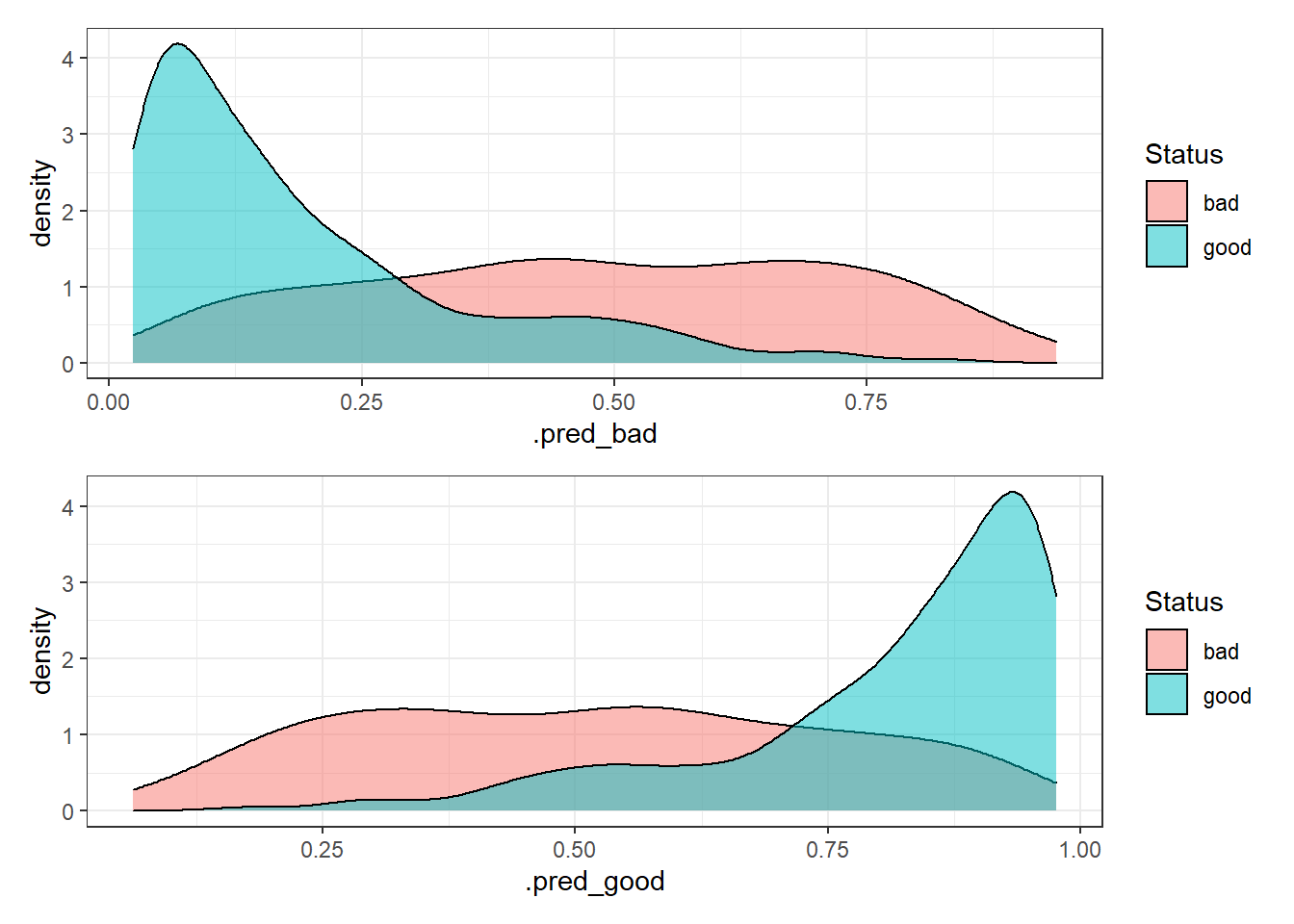
La performance du modèle XGboost n’est aussi pas optimale qu’attendue…Il faut revoir le process, peut-être en incrémentant d’autres données, revoir notre stratégie du tuning et du preprocessing, chnager le thresholds ou changer le modèle comme le Catboost ou LightGBM ; il y a toujours des pistes 😊
